USEARCH - 序列分析软件
USEARCH(Ultra-fast sequence analysis)是一款序列分析软件,在序列比对、聚类、操作等多领域被广泛的应用。在扩增子分析领域的OTU聚类非常受欢迎。该软件由Robert Edgar开发,在序列搜索、聚类、去重、去嵌合体等步骤的度以及效率上。
USEARCH的优点如下:
1.高通量搜索聚类
USEARCH是一个序列分析工具,拥有大量用户。UAEARCH比对速度是BLAST的10-1250倍,聚类速度是CD-HIT的1-1000倍。
2. 更高的生产率和洞察力
USEARCH将不同的算法结合到一个具有出色文档和支持的软件包中,这样可以缩短您的学习曲线,减少为给定任务所需采取的步骤,并减少计算时间。USEARCH会鼓励您探索数据,获得新见解,并建议您使用较慢的工具可能没有尝试过的新分析。
3. 安装简单便捷,程序小巧,易用
2018年7月底,USEARCH新版11已正式发布。新版Version 11,新增了6大新功能以及21个新命令。
新功能包含:
1.Machine learning:机器学习,主要包括随机森林、多次交叉验证、OTU分类相关性,包括的命令有森林训练、森林分类和森林交叉验证
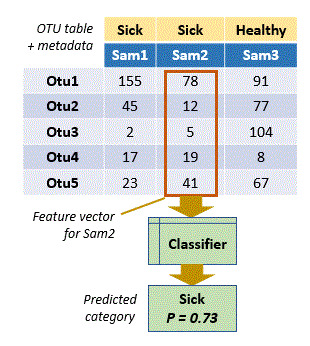
2.Improved cross-talk detection:改进嵌合体检测
3.Novel alpha diversity metrics:新增了两种Alpha多样性指数:Mirror estimator 、Singleton-free(FE)estimator
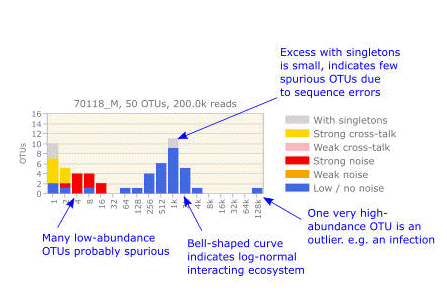
4.Octave plots for visualizing alpha diversity:Octave plots(八度图)展示Alpha 多样性,方便观察样品中真实序列、测序错误和嵌合体数量
5.Statistical significance of diversity differences between groups:样品组间多样性比较
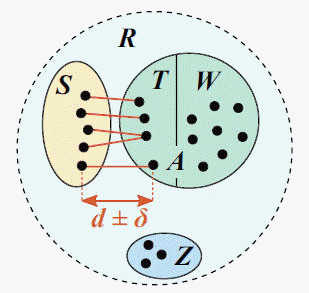
6.Cross-validation by identity (CVI):同一性交驻验证预测的率
新增命令:
- calc_lcr_probs:Calculate lowest common rank probabilities 计算序列物种分类各项概率
- cluster_tree:Construct clusters from tree using distance cutoff 基于树文件聚类
- distmx_split_identity:Split distance matrix into test/training pair for CVI 拆分距离矩阵为测试和训练集
- fastx_syncpairs:Sort forward and reverse reads into the same order 双端序列找成队并排序
- fastx_trim_primer:Remove primer-binding sequence from FAST×file 引物匹配并切除
- forest_classify:Classify data using random forest 随机森林分类预测
- forest_train:Train random forest classifier 随机森林分类器建立
- nbc_tax:Predict taxonomy using RDP Naive Bayesian Classifier algorithm 采用RDP算法预测分类学物种注释
- otutab_binary:Convert OTU table with counts to presence(1)/absence(0) 转换OTU表为二元(有、无)形式
- otutab_forest_classify:Classify samples using random forest 样品随机森林分类
- otutab_core:Identify core microbiome in OTU table 鉴定OTU表中核心微生物组
- otutab_forest_train:Train random forest classifier on OTU table 基于OTU表的随机森林训练
- otutab_otus:Extract OTU labels from OTU table 提取OTU表中的OTUs
- otutab_rare:Sub_sample OTU table to same number or reads per sample 抽样标准化OTU表
- otutab_samples:Extract sample labels from OTU table 提取OTUs表中样品名
- otutab_select:Identify OTUs which are informative (correlate with metadata)鉴定更有信息的OTUs,即组间差异OTUs
- otutab_xtalk:Identify cross-talk using improved algorithm(UNCROSS2)改进算法鉴定嵌合
- search_pcr2:In-silico PCR,search for matches to primer pair 电子PCR,基于引物匹配扩增区
- subtree:Extracts subtree under given node 提取树中指定结点的子树
- tabbed2otutab:Convert read mapping file (read+OTU)to OTU table 单行表格转换为OTU表
- tree_subset:Extract subset of tree for given set of leaf labels 根据树叶标签提取子集
- 2026-02-09
- 2026-01-20
- 2026-01-16
- 2026-01-12
- 2026-01-12
- 2026-01-09
- 2026-02-05
- 2026-02-05
- 2026-01-28
- 2026-01-26
- 2026-01-26
- 2026-01-16














